シリアルブロックフェイスイメージングとは
シリアルブロックフェイス走査電子顕微鏡法(SBEM、SBSEM、およびSBFSEM)は、試料から高解像度の三次元画像を再現可能な方法で取得する手法です。この手法は、ナノメートルレベルの分解能でX,Y,Zの広視野のイメージングを行う場合に特に適しています。通常、SBEMは、走査型電子顕微鏡(SEM)内部に組み込まれたin-situウルトラミクロトームを使用します。SEMが断面画像を収集すると、ウルトラミクロトームが15 nmという薄さで試料を切り出して、像観察を行う次の層を露出させます。
SBEMのメリット
| 機能 | メリット |
|---|---|
| 複数桁のスケールにわたり構造を観察 | 大きなデータセット全体にわたり詳細なデータを取得 |
| 操作不要で高スループットを実現 | 試料分析を高速化し、人為的なミスを軽減 |
| 観察された超微細構造の厚さ方向の関係を解明 | 1つの試料からより包括的な結果を取得 |
| セクショニングと像取得のあらゆるプロセスを管理 | 試料に対して条件を最適化するための完全な柔軟性を提供 |
| 切片の損失、損傷、変形の可能性を排除 | 損傷や変形を補正するために必要な処理を回避 |
用途
| 神経科学 | 細胞生物学 | 材料科学 | その他 |
|---|---|---|---|
|
|
|
|
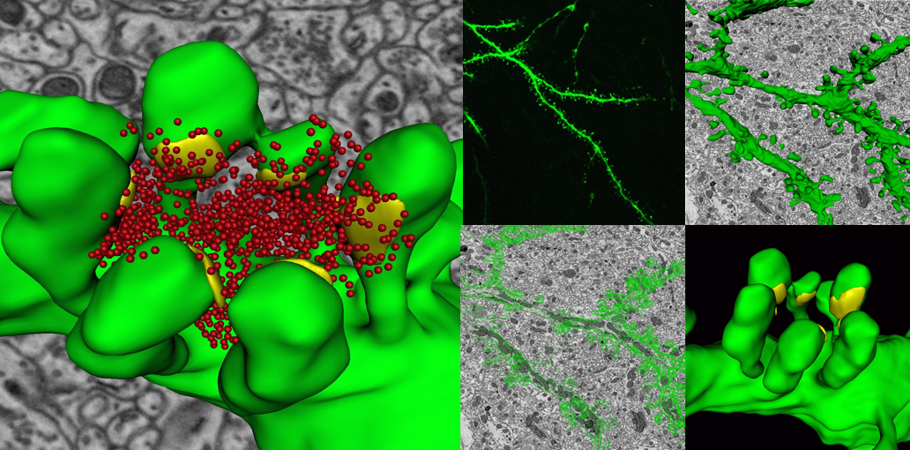
神経科学
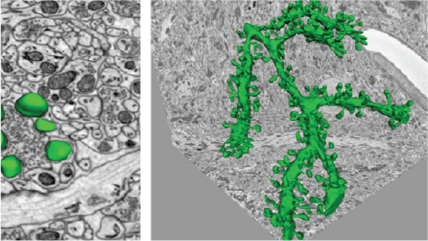
3View®システムにより生成されたマウス小脳の500枚の連続画像を含む、15,625 μm³ (25 x 25 x 25 μm)の三次元データセットを使用した樹状突起の三次元再構築像。樹状突起構造(緑色)、ボタン(黄色)、および小胞(赤色)。挿入画像は左上から時計回りに小脳の共焦点像、三次元モデルにレンダリングしたワイヤフレームトレース、超解像度の樹状突起棘モデルとシナプス、ワイヤフレームトレースを示す画像。
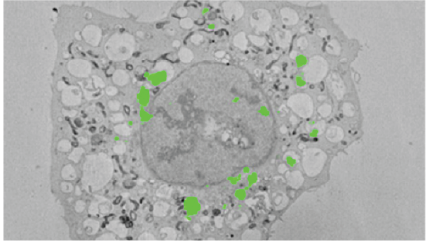
細胞生物学:共焦点の補完
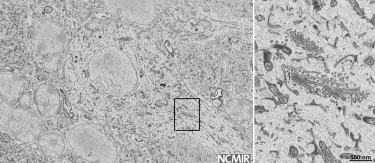
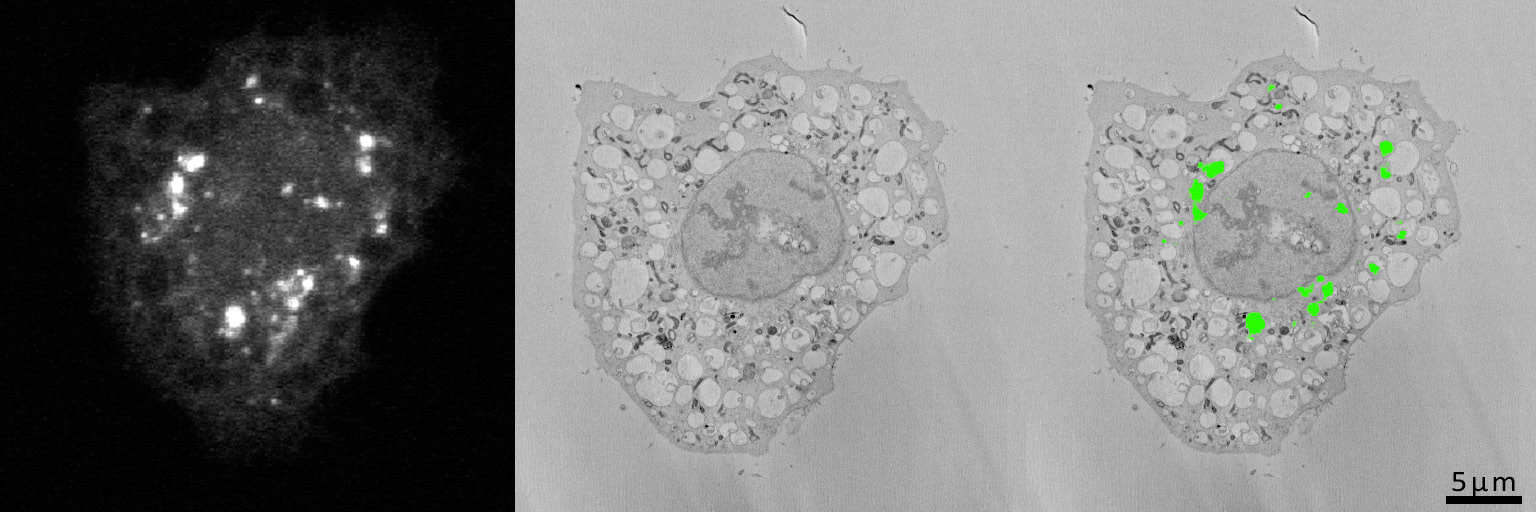
グリッド入りガラスボトムカバーガラスシャーレ上で培養されたLC-GFPを安定的に示しているヒーラ細胞の画像。無血清培地で2時間、飢餓状態にし、対象の細胞を共焦点顕微鏡法で識別したもの。細胞は電子顕微鏡用にin situで処理し、カバーガラスはフッ化水素酸を使用してエポキシ樹脂から溶解しています。細胞は樹脂ブロック内と、3Viewシステムで作成された連続画像内に観察されています。
細胞生物学:発生
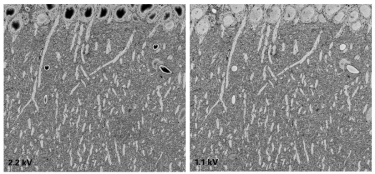
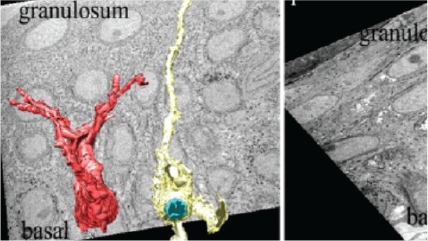
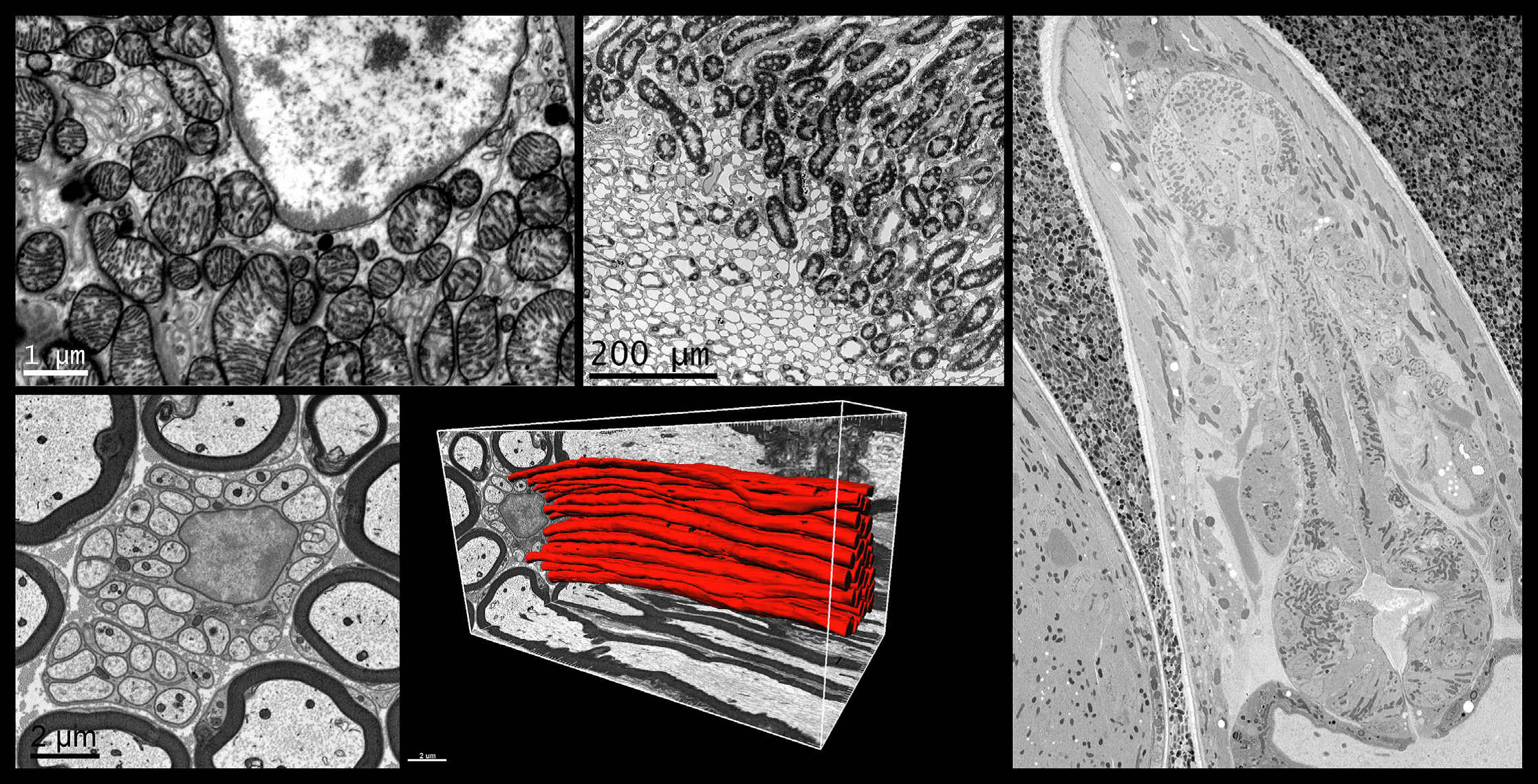
左上:高解像度、マウスの腎臓、1.5 nmピクセルで取得した8192 x 8192ピクセル画像。中央上:広視野、マウスの腎臓、80 nmピクセルで取得した8192 x 8192ピクセル画像。右:高圧凍結法で作製したC.エレガンス、25 nmピクセルで取得した4096 x 4096ピクセル画像。試料はカリフォルニア大学(バークレー)のKent McDonald様のご厚意によるものです。左下:マウスの坐骨神経、5 nmピクセルで取得した2048 x 2048ピクセル画像。中央下:マウスの坐骨神経軸索の三次元視覚化画像。
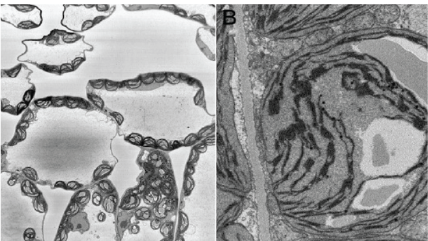
材料科学
左上および左下:3Viewシステムで作成したアルミニウム表面の陽極酸化被覆の画像。中央:3Viewシステムで作成したアルミニウム合金とマンガン粒子の三次元可視化画像。3Dデータセットには、ピクセルサイズ15 nm、切片厚さ15 nmの1024 x 1024連続画像1,000枚が含まれています。3Dモデルは、3D可視化プラグインを使用してDigitalMicrograph®で作成されたものです。

シリアルブロックフェイスイメージングのワークフロー
|
|
ステップ1:試料作製 通常、生物試料は固定し、染色液で染色し、安定に保持するため樹脂に埋め込みます。 |
 |
ステップ2:試料マウントとSEMへの移送 試料は必要な寸法に合わせてトリミングし、アルミニウムピンに載せて固定し、任意で金薄スパッタコーティングを行います。試料ピンを3Viewシステムに固定し、ダイヤモンドナイフを接触させ、ドアを閉めて排気します。 |
 |
ステップ3:イメージングの最適化 標準的なSEM条件に従い、ただし、Z方向の影響を考慮して、適切なビーム条件を選択します。画像の解像度、視野、取得時間が目的の条件になるように、倍率、ピクセル数、およびドウェルタイムを選択します。 |
 |
ステップ4:自動像観察 選択したビーム条件で連続画像が取得されます。各画像を取得する前に、ナイフで試料の表面層が切り取られます。 |
 |
ステップ5:解析 画像は三次元データセットを形成するためにスタックされ、このデータセットをDigitalMicrograph®またはサードパーティ製のソフトウェアで処理して表示できます。この時点で、セグメンテーションと定量化が可能になります。 |